
传统的实验方法主要包括 MNase-seq 和 DNase-seq,它们的主要思路是:当染色质变得开放时,DNA 和组蛋白的聚集程度降低,导致一部分 DNA 暴露出来。失去蛋白质的保护后,这部分 DNA 可被 DNA 酶(如 MNase 或 DNase I)切割。随后,我们对切割后的 DNA 进行测序,并将其与已知的全基因组序列进行比较,以确定哪些序列被切割,哪些基因序列未受影响,从而确定开放的染色质区域。但是这两种方法存在明显的缺陷,主要表现在耗时费力和重复性差的问题。还有 FAIRE-Seq,先进行超声裂解,然后用酚-氯仿富集,不依赖酶和抗体,但弊端就是检测背景高,测序信噪比低,甲醛交联时间不好把握等等。ChIP-Seq 是揭示特定转录因子或蛋白复合物的结合区域,实际是研究 DNA 和蛋白质的相互作用,利用抗体将蛋白质和 DNA 一起富集,并对富集的 DNA 测序。整体的分析思路一致,这些技术找富集区域,对富集区域进行功能分析。相比起来,ATAC-seq 是用Tn5 转座酶,操作起来也更加简单,重复性好,而且最重要的一点是实验只需要很少的细胞/组织量,出来的信号结果更好,所以 ATAC-seq 目前已经是研究染色质开放性首选的技术方法。
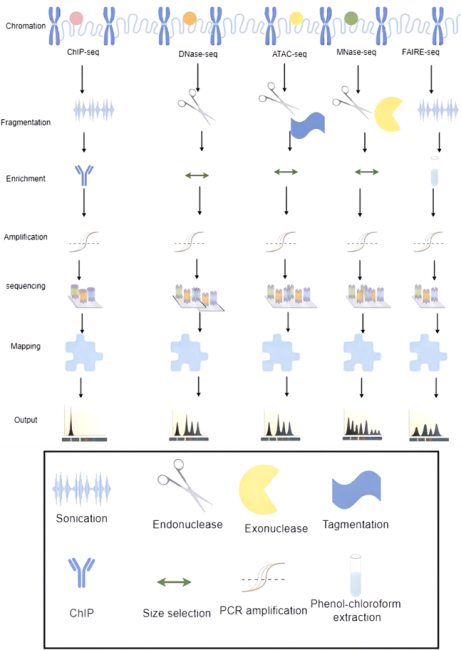
ATAC-seq(Assay for Transposase-Accessible Chromatin with high-throughput sequencing)是一种基于高通量测序的技术,用于研究染色质的可及性。
该技术的原理如下:
染色质结构: 在真核生物中,DNA并非裸露在细胞核中,而是与组蛋白结合,形成核小体结构。这些核小体进一步折叠和聚合,形成染色体。在基因表达过程中,DNA的高级结构会被部分解开,形成开放染色质区域,这些区域是转录因子和调控元件结合的关键位点。
实验过程:ATAC-seq利用Tn5转座酶,这种酶能够识别并进入细胞核中的开放染色质区域。Tn5酶能够在这些区域切割DNA,并在切割的同时将已知的DNA序列标签连接到DNA的两端。这些标签用于PCR扩增和随后的高通量测序。
数据分析: 通过生物信息学分析,研究人员可以确定全基因组范围内开放染色质区域的位置和特征。这些区域与转录因子结合位点、组蛋白修饰等关键生物学信息密切相关。
ATAC-seq技术因其高灵敏度和全基因组覆盖范围,已成为研究染色质可及性和转录调控的重要方法。
1.发育过程中染色质构象变化及其介导的基因表达变化;
2.特定生物学变化(如癌变)前后染色质构象变化及其介导的基因表达变化;
3.特定对照-处理前后染色质构象变化及其介导的基因表达变化;
4.研究非编码RNA在染色质构象调控中的功能;
5.鉴定特定生物学过程中的主效调控的转录因子;
6.新型增强子的鉴定;
ATAC-seq作为研究三大表观调控因素之一的手段,通常与组蛋白修饰、DNA修饰以及基因表达一起联合使用。
1.样本准备;
2.Tn5转座反应;
3.文库构建;
4.高通量测序;
5.数据分析。
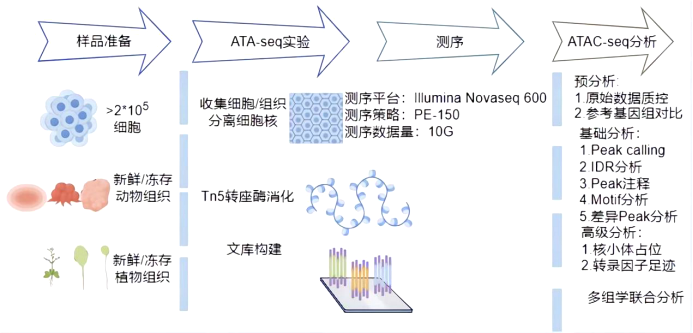
1. 全物种覆盖:独创全物种细胞核活性分离技术,彻底解决植物、微生物以及复杂组织无法进行ATAC-seq研究的难题,做到全物种(真核)、全组织类型兼容
2. 冷冻组织兼容:独家CL-ATAC-seq技术,实验前固定核小体结构,维持ATAC-seq过程中染色质构象,从而使得冷冻组织ATAC-seq研究成为可能。
3. 一站式服务:提供样品处理、ATAC建库、测序和分析的全套服务;
4. 项目经验丰富:已完成十多个物种、数十种组织类型的ATAC-seq服务。
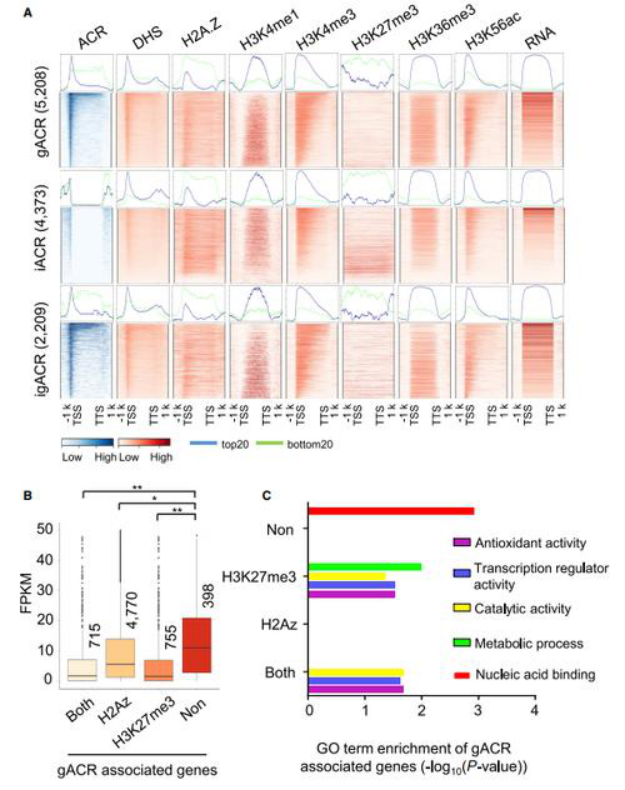
(Zhou et al., 2021, Plant Communications)