
| 项目编号 | demo |
|---|---|
| 项目名称 | 植物线粒体基因组组装 |
| 参考基因组 | Species name |
| 物种名称 | Species name |
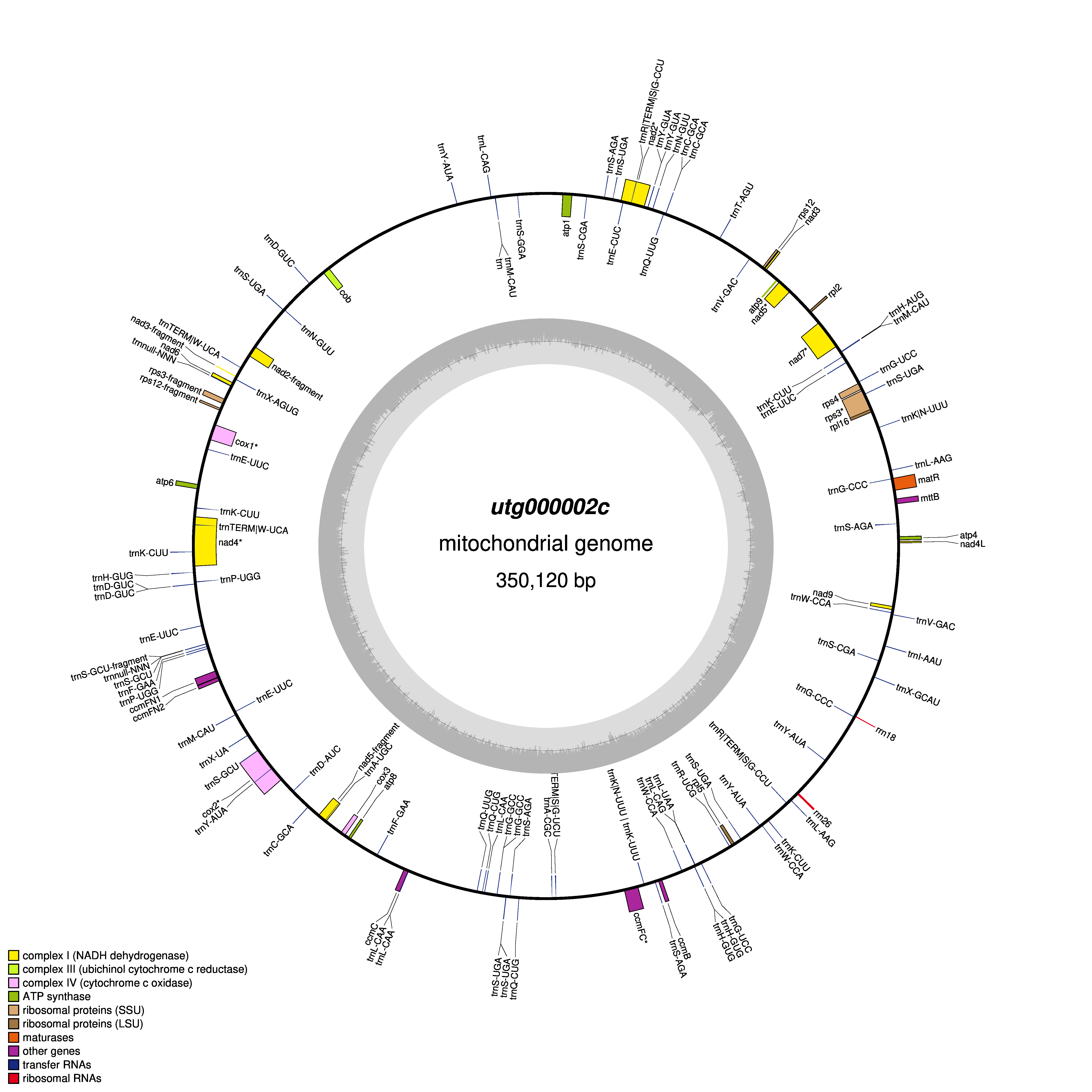
植物线粒体基因组的构象是多样的,组装出的线粒体基因组有可能是环形(大多数植物线粒体基因组),有可能是多环(玉米和猕猴桃线粒体基因组),有可能是线性(生菜线粒体基因组),有可能是多分支结构(北美云杉线粒体基因组),在多篇顶级期刊的论文中均证明了该观点。大多数情况下,植物线粒体基因组保持了单一环形的结构,但是也会由于部分重复序列介导重组溢裂成多个小环,这些情况都是真实存在的,例如茶树线粒体基因组,猕猴桃线粒体基因组等,因此无需担心结果为多个环形分子。在线性的植物线粒体基因组结构中,我们通过混合组装该线粒体基因组,可以恢复该线性分子的闭合多分支结构,从而证明其完整性,因此无需担心会线性基因组不完整从而影响发表。
(1)线粒体基因组的组装方法:我们使用 Illumina / BGI 的二代测序数据和 Nanopore / Pacbio 的三代测序数据,采用混合组装的策略组装线粒体基因组。使用 miniasm 软件对三代 DNA 测序数据进行植物线粒体组装,利用 racon 和 pilon 软件基于三代和二代测序数据进行基因组纠错,得到植物线粒体基因组。
(2)线粒体基因组的注释方法:线粒体基因组的蛋白质编码基因选取拟南芥(Arabidopsisthaliana, NC_037304)作为参考基因组,使用 Geseq 软件对线粒体基因组的基因组进行注释。线粒体基因组的 tRNA 和 rRNA 均在该软件被注释注释。每一个线粒体基因组的注释错误都使用 Apollo 软件手动修改纠错。
| Species name | Contigs | Type | Length | GC content |
|---|---|---|---|---|
| Arabidopsis thaliana | Chromosome | circular | 350,120 bp | 45.26 % |

| Groups of genes | Name of genes |
|---|---|
| ATP synthase | atp1,atp4,atp6,atp8,atp9 |
| NADH dehydrogenase | nad1,nad2,nad3,nad4,nad4L,nad5,nad6,nad7,nad9 |
| Cytochrome c biogenesis | cob |
| Ubiquinol cytochrome c reductase | ccmB,ccmC,ccmFC,ccmFN1,ccmFN2 |
| Cytochrome c oxidase | cox1,cox2,cox3 |
| Maturases | matR |
| Transport membrane protein | mttB |
| Large subunit ofribosome | rpl16,rpl2,rpl5 |
| Small subunit ofribosome | rps3,rps4,rps12 |
| Succinate dehydrogenase | |
| Ribosome RNA | rrn18,rrn26 |
| Transfer RNA | trnA-CGC,trnA-UGC,trnC-GCA (3),trnD-AUC,trnD-GUC (3),trnE-CUC,trnE-UUC (4),trnF-GAA (2),trnG-CCC (2),trnG-GCC (2),trnG-UCC (2),trnH-AUG,trnH-GUG (3),trnI-AAU,trnK-CUU (4),trnK-UUU,trnL-AAG (2),trnL-CAA (3),trnL-CAG (2),trnL-UAA,trnM-CAU (3),trnN-GUU (3),trnP-UG (2),trnQ-CUG (2),trnQ-UUG (2),trnR-UCG,trnS-AGA (4),trnS-CGA (2),trnS-GCU (2),trnS-GGA,trnS-UGA (6),trnT-AGU,trnV-GAC (2),trnW-CCA (3),trnX-AGUG,trnX-GCAU,trnX-UA,trnY-AUA (4),trnY-GUA (2),,trnnull-NNN (2),trnK|N-UUU | trnK-UUU,trnM-CAU | tRNAI(CAT),trnR|TERM|S|G-CCU (2),trnR|TERM|S|G-UCU,trnTERM|W-UCA (2) |
该物种线粒体基因组总长度为 350,120 bp,GC 含量为 45.26 %。对其的线粒体基因组进行注释,共注释到 72 个 unique 蛋白质编码基因,40 个 tRNA 基因(其中 26 个 tRNA 为多拷贝),2 个 rRNA 基因。核心基因中包括 5 个 ATP合酶基因(atp1,atp4,atp6,atp8 和 atp9);9 个 NADH 脱氢酶基因(nad1,nad2,nad3,nad4,nad4L,nad5,nad6,nad7 和 nad9);5 个细胞色素 C 生物发生基因(ccmB,ccmC, ccmFC,ccmFN1和ccmFN2);3 个细胞色素 C 氧化酶基因(cox1,cox2 和 cox3);1 个膜运输蛋白基因(mttB);1 个成熟酶基因(matR)和 1 个泛醇-细胞色素C 还原酶基因(cob)。非核心基因包括 3 个核糖体大亚基基因(rpl16,rpl2,rpl5);3 个核糖体小亚基基因(rps3,rps4,rps12)。
真核生物基因组存在 64 个密码子,这 64 个密码子编码 20 种不同的氨基酸和 3 个终止密码子,除蛋氨酸和色氨酸外的所有氨基酸均由多个密码子编码。不同物种、不同生物体的基因组密码子使用率存在着较大的差异。这种偏好性被认为是在长期的演化选择中,细胞内逐渐形成相对平衡的结果。因此,在基因组分析中通常对密码子偏好性进行分析(Relative Synonymous Codon Usage, RSCU)。
使用 python 脚本提取基因组的蛋白质编码序列。使用 python脚本对线粒体基因组的蛋白质编码基因(筛选长度大于300bp且为3的倍数的PCG)进行密码子偏好性分析,并计算 RSCU 值。
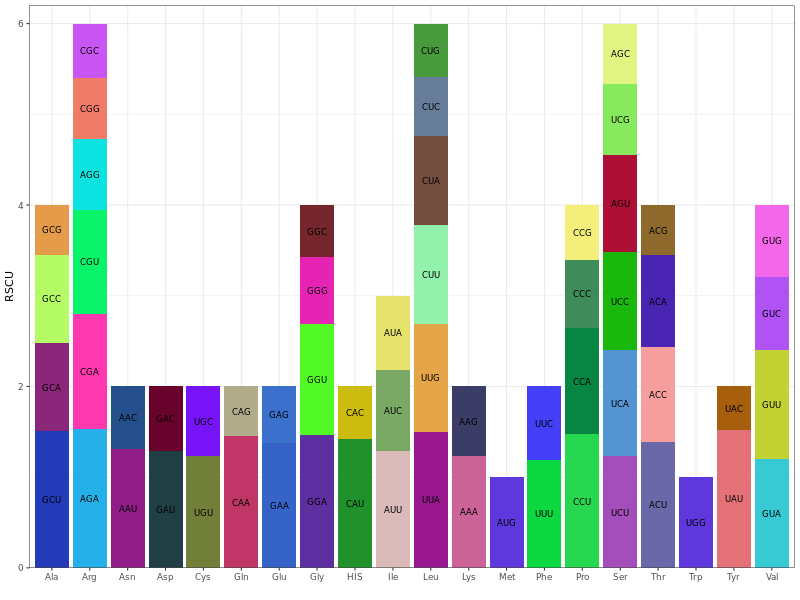

对该物种线粒体的 19 个 unique PCGs 进行密码子偏好性分析,各个氨基酸对密码子的使用情况见表 3-1。相对同义密码子使用度(RSCU)大于 1 的密码子被认为是被氨基酸偏好性使用的。如图 3-1 所示,除甲硫氨酸(Met-ATG)和色氨酸(Trp-TGG)的 RSCU 值均为 1 之外,其他线粒体的 PCGs 均存在普遍的密码子使用偏好。
| Amino | Codon 1 RSCU | Codon 2 RSCU | Codon 3 RSCU | Codon 4 RSCU | Codon 5 RSCU | Codon 6 RSCU |
|---|---|---|---|---|---|---|
| Ala | GCA 0.98 | GCC 0.97 | GCG 0.55 | GCT 1.50 | ||
| Arg | AGA 1.53 | AGG 0.78 | CGA 1.27 | CGC 0.60 | CGG 0.67 | CGT 1.15 |
| Asn | AAC 0.69 | AAT 1.31 | ||||
| Asp | GAC 0.71 | GAT 1.29 | ||||
| Cys | TGC 0.77 | TGT 1.23 | ||||
| Gln | CAA 1.45 | CAG 0.55 | ||||
| Glu | GAA 1.38 | GAG 0.62 | ||||
| Gly | GGA 1.46 | GGC 0.57 | GGG 0.74 | GGT 1.23 | ||
| His | CAC 0.58 | CAT 1.42 | ||||
| Ile | ATA 0.82 | ATC 0.89 | ATT 1.29 | |||
| Leu | CTA 0.98 | CTC 0.65 | CTG 0.59 | CTT 1.08 | TTA 1.50 | TTG 1.20 |
| Lys | AAA 1.23 | AAG 0.77 | 0.82 | |||
| Met | ATG 1.00 | |||||
| Phe | TTC 0.81 | TTT 1.19 | ||||
| Pro | CCA 1.17 | CCC 0.75 | CCG 0.61 | CCT 1.47 | ||
| Ser | AGC 0.66 | AGT 1.06 | TCA 1.17 | TCC 1.09 | TCG 0.79 | TCT 1.23 |
| Thr | ACA 1.01 | ACC 1.04 | ACG 0.56 | ACT 1.39 | ||
| Trp | TGG 1.00 | |||||
| Tyr | TAC 0.48 | TAT 1.52 | ||||
| Val | GTA 1.21 | GTC 0.81 | GTG 0.79 | GTT 1.19 |
生物细胞中的 DNA 序列里面包含许多重复序列(Repeated sequence),主要可分为两大类,分别是串联重复序列(也叫串接重复序列,Tandem repeat)与散在重复序列(Dispersedrepeat)。串联重复和散在重复的区别在于重复的部分是否相邻分布,相邻就是串联重复,不相邻就是散在重复。在串联重复中,微卫星重复序列(Simple Sequence Repeat, SSR)是一种特殊的串接重复序列,一般不超过 6 bp。由于 SSR 具有显性遗传等特点,经常被用于分子标记开发。
分别用 MISA 软件、TRF 软件和 REPuter 软件识别了包括微卫星序列重复、串联重复和散在重复的重复序列。使用 R 软件对结果进行了可视化。

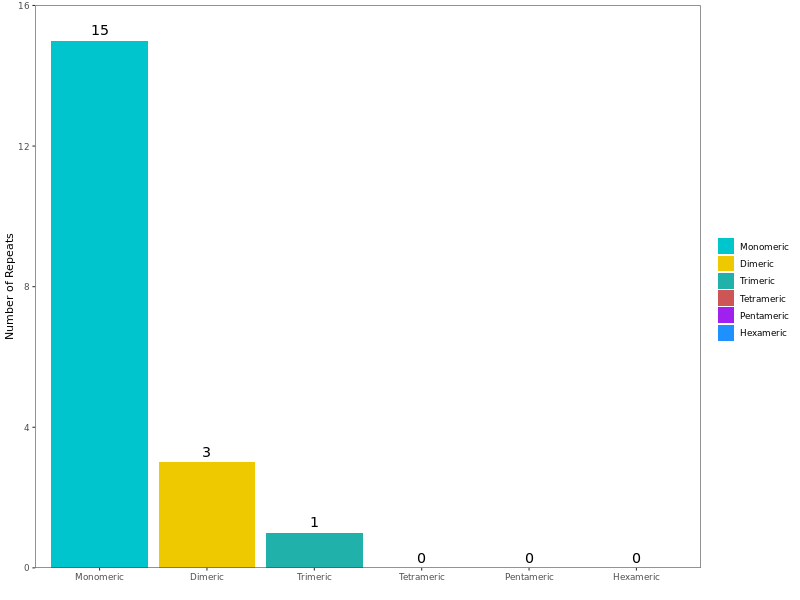
图 3-1-1: The number of repeats for different types of SSRs
横坐标表示不同类型SSR,纵坐标表示重复片段数量,Monomeric表示单体 SSRs,Dimeric表示二聚体 SSRs,Trimeric表示三聚体 SSRs,Tetrameric表示四聚体 SSRs,Pentameric表示五聚体 SSRs,Hexameric表示六聚体 SSRs。
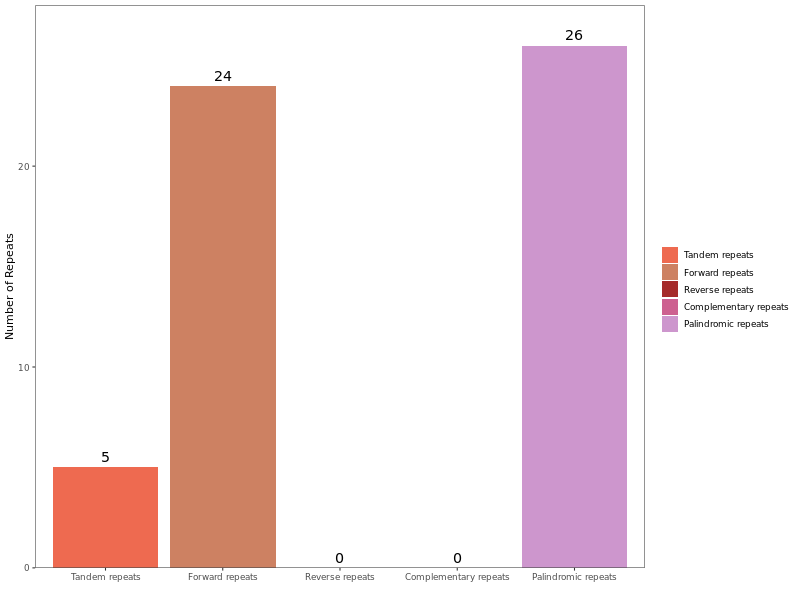

横坐标表示不同类型的TR和DR,纵坐标表示重复片段数量,串联重复:Tandem repeats,回文重复:Palindromic repeats,正向重复:Forward repeats,反向重复:Reverse repeats,互补重复:Complementary repeats。
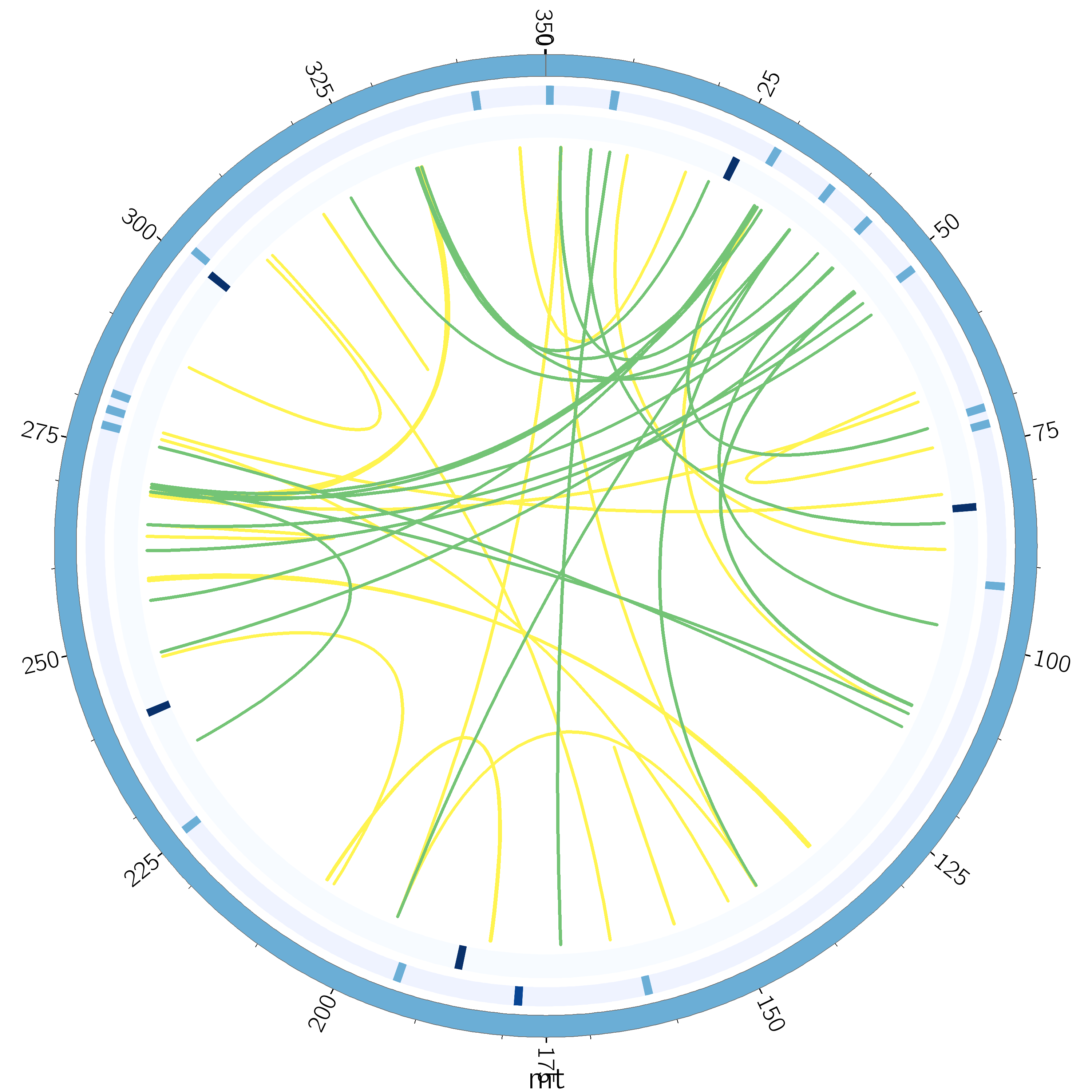
对线粒体基因组分别进行重复序列分析,最里面的圆圈上的彩色线条连接了分散重复的两个重复序列,绿色线条代表回文重复(Palindromic repeats),黄色线条代表正向重复(Forward repeats),蓝色线条代表反向重复(Reverse repeats),红色线条代表互补重复(Complementary repeats)。第二个圆圈上的深蓝色线段代表串联重复序列,最外侧的圆环上的浅蓝色线段代表微卫星重复序列。
在线粒体基因组中共发现 19 个 SSRs,单体和二聚体形式的 SSRs 占总 SSRs 的 78.95 %,未发现四聚体、五聚体以及六聚体 SSRs。串联重复序列,又称卫星 DNA,是指 7~ 200 个碱基左右的核心重复单元,以串联方式重复多次。它们广泛存在于真核生物基因组和原核生物中。线粒体基因组中共有 5 个匹配度大于 75 %,长度在 15~37 bp 之间的串联重复序列。对散在重复序列进行了检测。结果共观察到长度大于或等于 30 的重复序列 50 对,其中回文重复(Palindromic repeats)26 对,正向重复(Forward repeats)24 对,没有检测到互补重复(Complementary repeats)和反向重复(Reverse repeats)。最长的回文重复为 6590 bp,最长 的正向重复为 4193 bp。
在“1.1 植物线粒体基因组的结构简介”部分,我们介绍了线粒体基因组可能的构象,例如环形、线性和多分支。以环形为例,某植物线粒体基因组主要构象为单一环形,由于线粒体基因组有大量的重排和重组的现象,因此它的结构可能不仅仅是单一的环形结构,还存在其他的亚结构。
使用 miniasm 软件组装图形化线粒体基因组,使用的参数为默认参数。

我们使用 Bandage 软件可视化该线粒体基因组草图,最终结果如图 4-1 所示。
| Contigs | Type | Path |
|---|---|---|
| 1 | circular |
在线粒体进化的过程中,部分叶绿体片段会迁移到线粒体基因组中,迁移片段的长度和序列相似性在不同物种之间不同。
使用 miniasm 软件对叶绿体基因组进行组装,使用 Geseq 软件对叶绿体基因组进行注释,然后使用 Apollo 软件对叶绿体基因组的注释结果进行矫正。通过使用 blast 软件对同源片段进行分析,使用 circos 软件对结果进行了可视化。
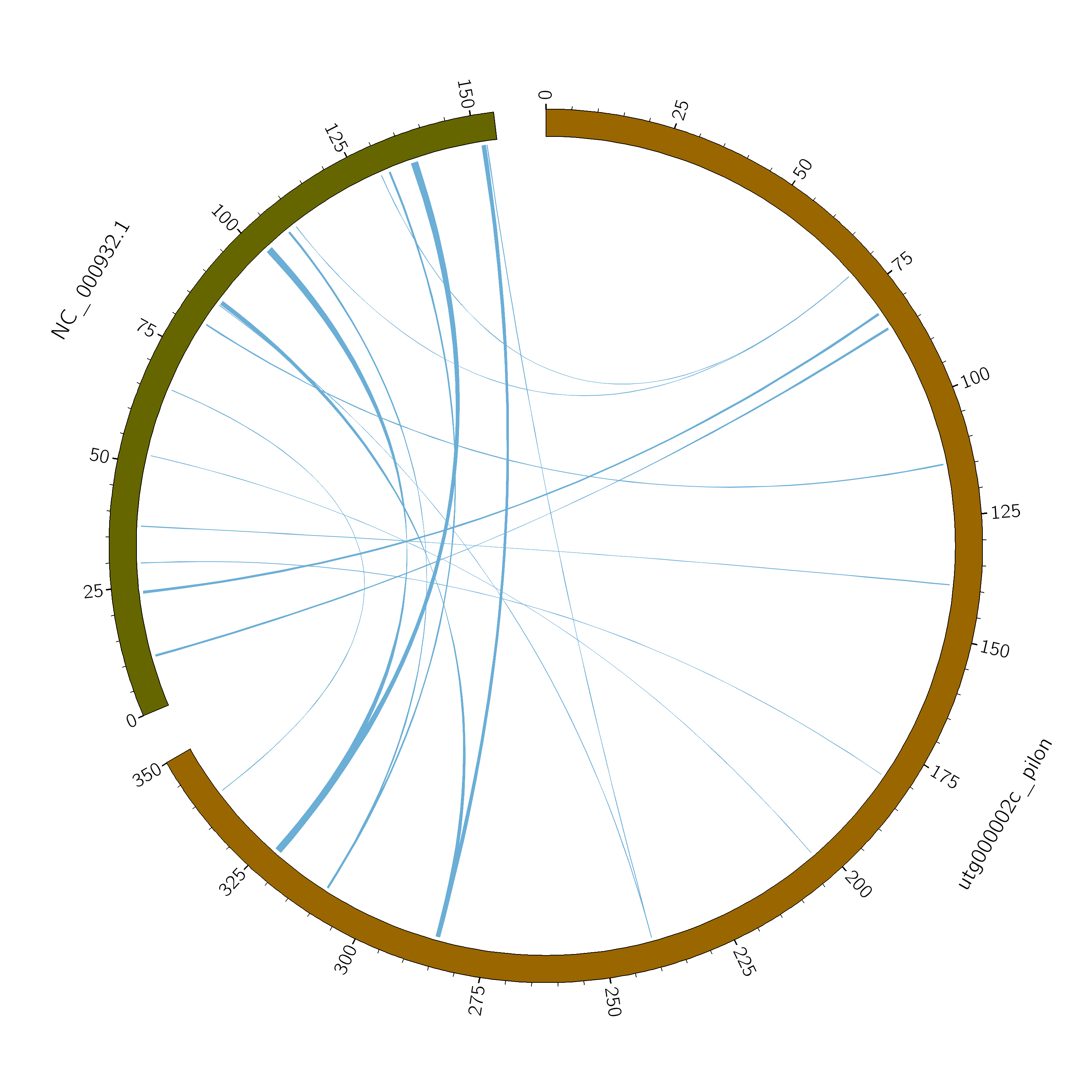

根据序列相似性分析,一共有 17 个片段为线粒体基因组和叶绿体基因组的同源片段,总长度为 7145 bp,占线粒体基因组总长的 2.04 %。其中 MTPT1、MTPT2 最长,为 1407 bp。
利用植物线粒体 DNA 分子(通常为保守的线粒体蛋白质基因的编码序列)数据重建系统发育树推断系统发育过程,分析物种之间的进化关系。通常用系统发育树表示。
根据亲缘关系,选择近缘物种并下载它们的线粒体基因组(根据该项目物种选择),然后使用 python 脚本提取共有基因,使用 mafft 软件进行多重序列比对分析,然后使用 iqtree 软件进行系统发育分析,使用R软件对系统发育分析的结果可视化。
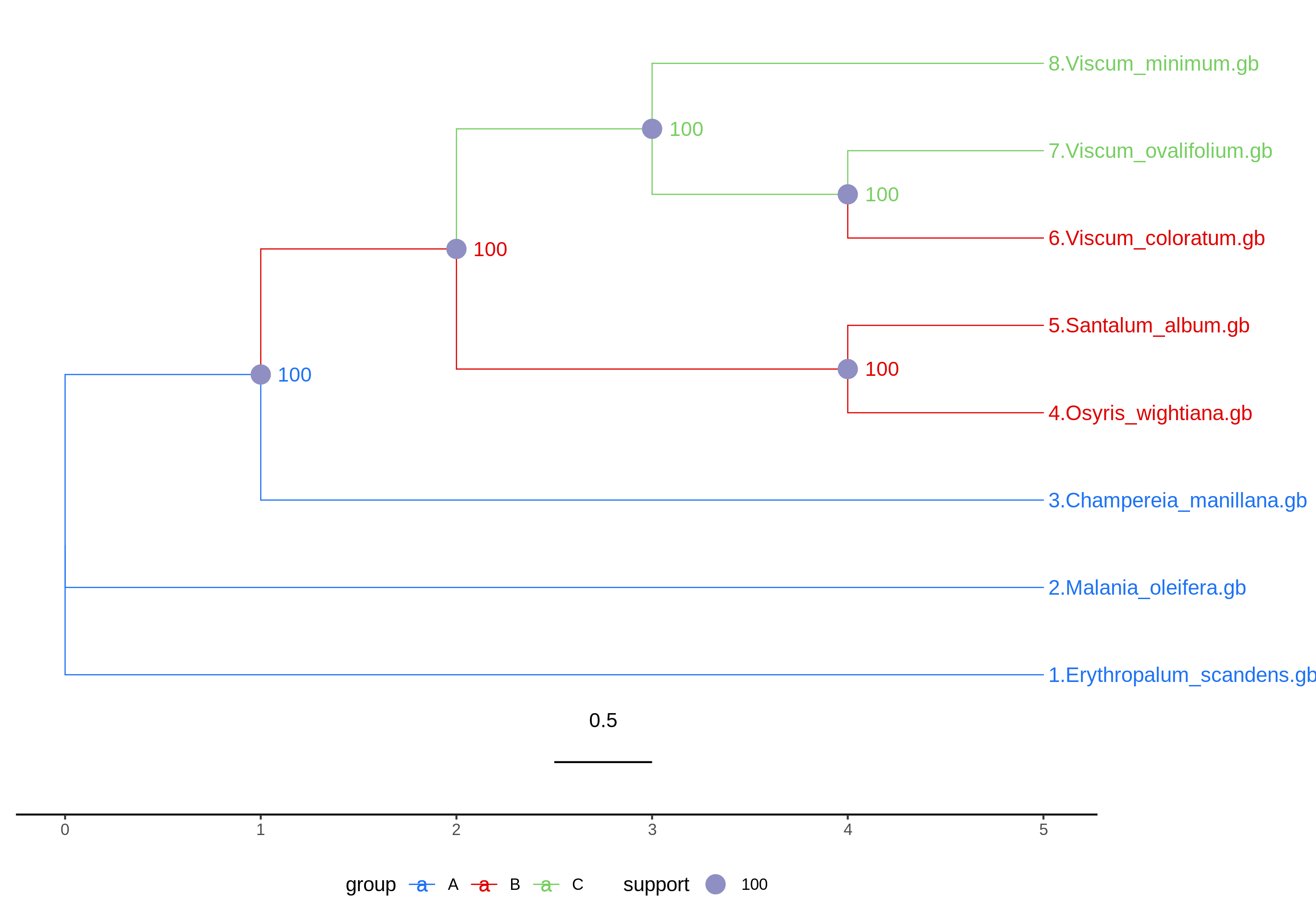

(仅描述信息,无实际意义)
基于 XXX 个保守的线粒体蛋白质编码基因(Protein Coding Genes, PCGs)的 DNA 序列,对被子植物 XX 个目下的 XXX 个物种进行了系统发育树的构建,具体植物物种线粒体基因组序列见附件 1。共有蛋白质编码基因分别为 atp4,atp6,ccmB,ccmC,ccmFC,cob,cox1,cox2,cox3,nad1,nad2,nad4,nad5,nad6,nad7,rpl16,rps3,rps4,rps7,rps12,rps13,sdh4。XXX 目的 2 个线粒体基因组被设置为外类群。基于线粒体 DNA 的系统发育的拓扑结构与 APG(Angiosperm Phylogeny Group)最新的分类相吻合。物种 XXX 属于 XXX 目 XXX 科,与 XXX 的亲缘关系较近。
共线性研究的是同源基因或者序列的排布关系,比较常见的是组装基因组文章中,通过同源性比较来评估基因组的组装效果以及同源基因的保留和丢失的情况,用来研究材料的进化关系。
基于 XXX 程序获得各个线粒体基因组两两比较的结果,并保留了长度超过 500 bp 的同源序列作为保守的共线块绘制 Multiple Synteny Plot。
基于序列相似性,我们使用 XXX 软件的源程序绘制了 XXX 与近缘物种的 Multiple Synteny Plot,结果如下:
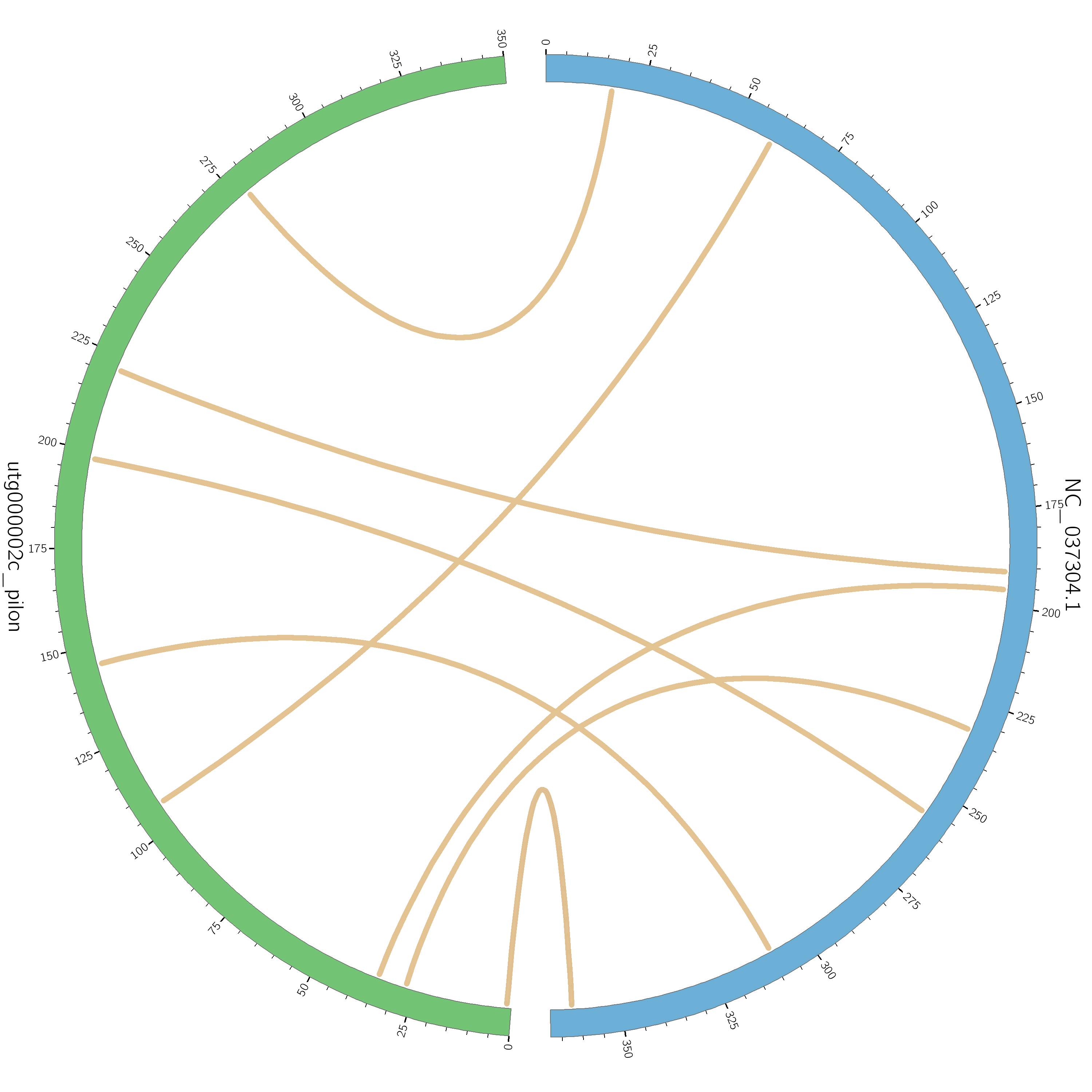

如图 7-1 所示,黄色线条区域表示同源性良好的区域。XXX 物种与 XXX 物种检测到了大量同源的共线块。此外,还发现一些空白的区域,这些序列是该物种中独有的,与其余物种没有同源性。结果表明,XXX 目各个线粒体基因组之间的共线块排列顺序不一致,XXX 线粒体基因组与近缘物种之间经历了大量的基因组重排,共线块长度较短,表明 XXX 目这 XX 个物种的线粒体基因组序列在排列顺序上极度不保守,经历了极其频繁的基因组重组。
1989 年,来自加拿大、法国、德国的三个实验室几乎同时独立报道了植物线粒体 RNA 中 C-U 编辑这一现象 后来的研究发现,RNA 编辑在高等植物线粒体中广泛存在,它是植物线粒体基因组中基因表达的必需步骤之一。RNA 编辑属于转录后修饰范畴,其化学本质是脱氨基反应,在这个过程中,一个特异位点的胞嘧啶(C)被改变成尿嘧啶(U)这种 C-U RNA 编辑 事件倾向于出现在第二个密码子位置,并且大多数都是完全编辑,通过编辑,提高了不同物种之间线粒体蛋白质序列的同源性。RNA 编辑位点中 92%会改变氨基酸序列,通常是将亲水性氨基酸转变成疏水性氨基酸,使蛋白质能更好地折叠并发挥功能。RNA 编辑还可以产生在基因组序列中所不存在的起始密码子和终止密码子,而通常产生新的起始密码子和终止密码子后,其编码的蛋白质更加保守,与其他物种相应蛋白质的同源性更高,从而使线粒体中的基因可以更好地进行表达。
基于 PREPACT 软件对 RNA 编辑事件进行预测(未提供转录组数据)。
考虑到我们没有转录组数据,因此,基于 PREPACT 软件对 RNA 编辑事件进行预测,共预测到了 413 个 RNA 编辑位点,其中348个是 C-T(U)的编辑,65个是U-C的编辑。各个基因中预测的 RNA 编辑位点如图 8-1 所示。
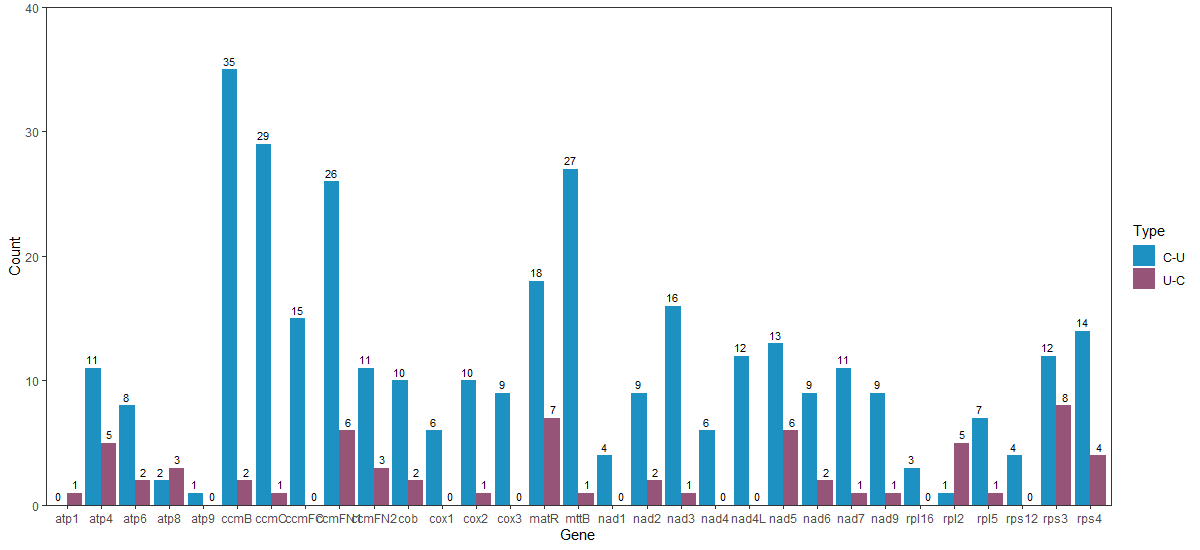

基于 PREPACT 软件预测,对来自线粒体的 31 个 unique PCGs 的 RNA 编辑事件进行了鉴定。设置标准为:cutoff value = 0.001。在这一标准下,在 31 个线粒体 PCGs 上共鉴定了 413 个潜在的 RNA 编辑位点,其中 348 个是 C-T(U)的编辑,65 个是 U-C 的编辑。在线粒体基因中,ccmB 基因鉴定了 37 个 RNA 编辑位点,在线粒体全部基因中,它的编辑次数最多。其次是 ccmFC 基因,发生了 30 次 RNA 编辑事件。其中 atp1 基因没有发现 C-U 的编辑,atp9、ccmFC、cox1、cox3、nad1、nad4、nad4L、rpl16 以及 rps12 没有发现 U-C 的编辑。